FSTAT & PCAGEN
ダウンロードはこちら→http://www2.unil.ch/popgen/softwares/
FSTATとPCAGENのinput fileはまったく同じ。GENEPOPのファイル変換(option7)を利用して作成できる。
sub-optionの1を選択してから、GENEPOPのinput fileを入力すると以下のようなResultsが出力される。
3 4 6 2 Loc1 Loc2 Loc3 Loc4 1 0202 0101 0305 0203 1 0104 0101 0205 0404 1 0202 0102 0103 0204 2 0204 0101 0405 0404 2 0102 0000 0103 0404 2 0202 0102 0102 0205 2 0205 0101 0305 0404 3 0204 0102 0103 0102 3 0206 0101 0303 0203 3 0203 0202 0203 0204 |
# 個体群の数 Locus数 最大Allele数 digit(1 or 2) # Locus名 # ここから個体データ 1列目の数字は個体群番号。 |
これをメモ帳などのテキストエディタに入力し、DATファイル形式[ .dat ]で保存する。(例:[test.dat])
現在作成中...
使い方は、[File]→[Open]から、DAT形式のinput fileを読み込んで、[Go]をクリックするだけ。
outputされるグラフを見やすくするために、[Go]の前に[Options]→[Label file for pops]から以下のようなファイルで個体群名を定義しておくといいかも。
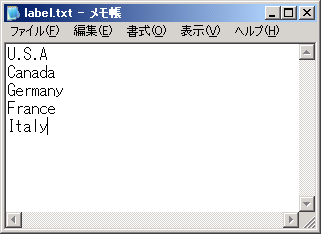
個体群名は任意。このようなファイルをテキスト形式などで作成する。
PCAGENがやってること。
PCAGENのinput fileのままでは主成分分析はできない。
PCAGENではこのファイルを、主成分分析ができる形に変換してるわけだが、
その変換方法をここで説明する。
PCAGENを使わなくても主成分分析ができるように、また、発表でつっこまれたときのためにも、 何を説明変数として主成分分析を行っているのかは、ぜひとも知っておきたい。
方法は、全ての対立遺伝子の頻度を、各個体群ごとに求めてるだけ。
(そしてこれはGENEPOPなどで簡単に計算できる。)
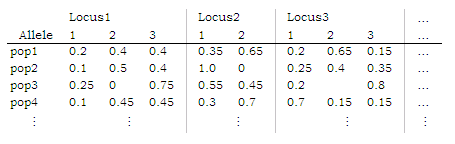
PCAGENでは、これをデータファイルとして主成分分析を行っている。
つまり、(全遺伝子座トータルでの)全対立遺伝子が主成分分析での説明変数となる。
統計ソフトRでのデータテーブルの例:
L1.1 L1.2 L1.3 L2.1 L2.2 L3.1 L3.2 L3.3 pop1 0.2 0.4 0.4 0.35 0.65 0.2 0.65 0.15 pop2 0.1 0.5 0.4 1.0 0 0.25 0.4 0.35 pop3 0.25 0 0.75 0.55 0.45 0.2 0 0.8 pop4 0.1 0.45 0.45 0.3 0.7 0.7 0.15 0.15