GENEPOP
ダウンロード不要。ウェブ上で使用可→Genepop on the Web
input fileはテキストファイルなどで以下のようにつくる。
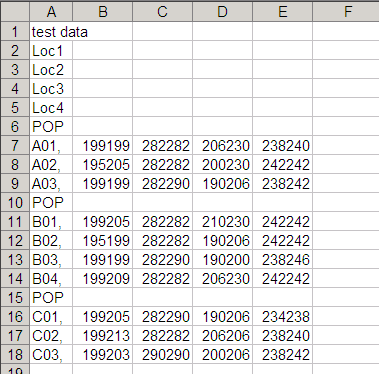
test data Loc1 Loc2 Loc3 Loc4 POP A01, 199199 282282 206230 238240 A02, 195205 282282 200230 242242 A03, 199199 282290 190206 238242 POP B01, 199205 282282 210230 242242 B02, 195199 000000 190206 242242 B03, 199199 282290 190200 238246 B04, 199209 282282 206230 242242 POP C01, 199205 282290 190206 234238 C02, 199213 282282 206206 238240 C03, 199203 290290 200206 238242 |
# タイトルなど自由に。 # Locusの名前。 「Loc1, Loc2, Loc3, Loc4」というように一行にしてもいい。 # 個体群の区切りにPOP(Pop, pop)と記入。 # ここから個体データ。IDとalleleの間にはコンマ。 # missing dataは「000000」 |
エクセル上で以下のように作られているなら、Excel関数を利用して比較的ラクにinput fileがつくれる。
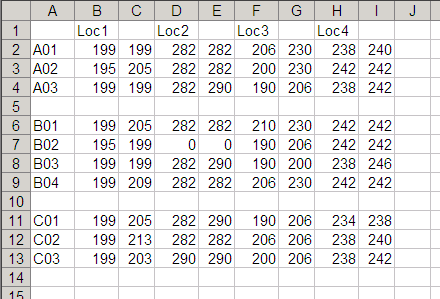
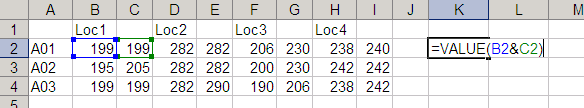
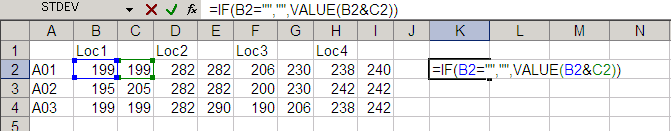
例えばセルK2に「=B2&C2」と入力すると、K2にはB2とC2の内容がくっついて「199199」と返される。
ここでは、値を数値化するために「=value(B2&C2)」と入力する(この方がたぶん後でラクになる)。
↓
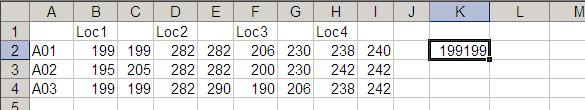
ここでK2とL2の2セルを選択して右へオートフィルでコピー。そしてそのまま下へもコピー。
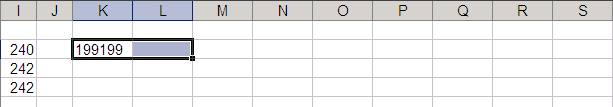
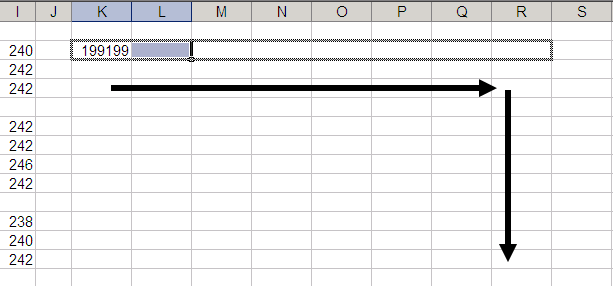
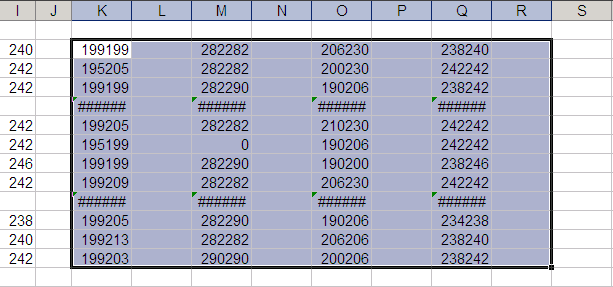
あとは、空白の列(L, N, P)を削除すればいい。
参照セルが空白のところは、「#######」のようにエラーになる。ここだけ後で消せばいいんだけど、こうなるのが面倒なら、一番最初に入力した関数を「=value(B2&C2)」ではなく、「=IF(B2="","",value(B2&C2)」と入力すれば大丈夫。
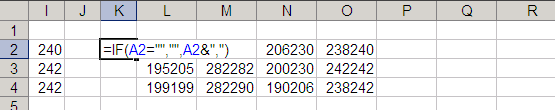
あと必要なのは、個体IDのあとのコンマ。 J列とK列の間に新しく列を挿入し、K2に「=IF(A2="","",A2&",")」と入力。あとはそれを下へコピーすればいい。
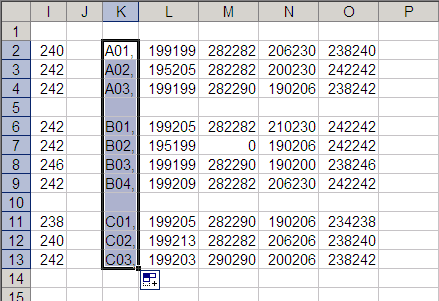
最後に、missing dataはalleleが3桁なら「000000」にしなきゃいけないが、これは、新しくできた表全体を選択して右クリックで「セルの書式設定」を選び、表示形式の分類で「ユーザー定義」を選び、右のボックスに「000000」と入力してOKすればいい。
これでほぼ完成!
あとは、最初に見せた完成版を見ながら、タイトル、locus名、個体群区切りの「POP」などを追加すれば完成。
テキストファイルなどに保存しとくと便利やけど、このExcelデータから直接コピペでGENEPOPに貼り付けても使える。
FSTATでもできるけど、たしかFSTATでは頻度(少数)で出力される。
Genepopでは、対立遺伝子数(整数)なので見やすい。
Genetic differentiation (Fisher's exact test)
option3option6
計算方法はFSTATと同じ。