STRUCTURE
ダウンロードはこちら→http://pritch.bsd.uchicago.edu/structure.html
ダウンロード(106KB)
アウトプットの対数尤度LnP(D)の値からΔKとかを簡単に求められます。
ただし、繰り返し数(Number of Iterations)を「10」にした場合の結果に限ります。
いじったら変更できるかもしれませんが。
input fileは以下のようにつくる。
保存形式は特に指定されてないけど、Excelでつくってタブ区切りでテキスト形式で出力したもの(拡張子[ .txt ])でいいと思う。
Loc1 Loc2 Loc3 Loc4 A01 1 199 282 206 238 A01 1 199 282 230 240 A02 1 195 282 200 242 A02 1 205 282 230 242 A03 1 199 282 190 238 A03 1 199 290 206 242 B01 2 199 282 210 242 B01 2 205 282 230 242 B02 2 195 -9 190 242 B02 2 199 -9 206 242 B03 2 199 282 190 238 B03 2 199 290 200 246 B04 2 199 282 206 242 B04 2 209 282 230 242 C01 3 199 282 190 234 C01 3 205 290 206 238 C02 3 199 282 206 238 C02 3 213 282 206 240 C03 3 199 290 200 238 C03 3 203 290 206 242 |
# 1行目はmarker name(Locus名)。(この行はなくてもよい) # 2行目以降は個体データ。 1列目は個体番号、2列目は個体群番号。この2列はなくてもよい。 # missing dataは「-9」(なぜかこの値が推奨) |
上の例では、二倍体のときのAlleleは縦並びになってるけど、以下のような横並びでもよい。
ソフトのヘルプファイルでは縦並びで説明されてるけど、横並びの方が作りやすいと思う。
Loc1 Loc2 Loc3 Loc4 A01 1 199 199 282 282 206 230 238 240 A02 1 195 205 282 282 200 230 242 242 A03 1 199 199 282 290 190 206 238 242 B01 2 199 205 282 282 210 230 242 242 B02 2 195 199 -9 -9 190 206 242 242 B03 2 199 199 282 290 190 200 238 246 B04 2 199 209 282 282 206 230 242 242 C01 3 199 205 282 290 190 206 234 238 C02 3 199 213 282 282 206 206 238 240 C03 3 199 203 290 290 200 206 238 242 |
input fileをSTRUCTUREに読み込むには、[File]→[New Project]を開く。
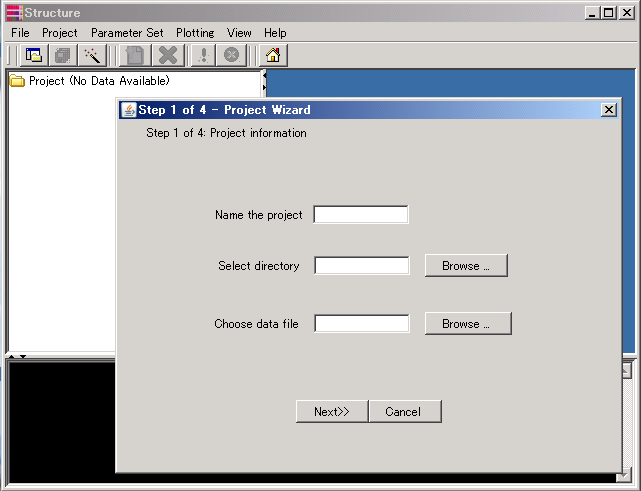
| Name the project | :タイトル。自由。 |
| Select directory | :結果ファイルを置くフォルダ(通常はinput fileのあるフォルダでよい)を指定する。 |
| Choose data file | :input fileを読み込む。 |
[Next>>]をクリックして、順次、データの情報を入力していく。一枚目以降はたぶんそんなに難しくないはず。
総個体数やローカス数を入力するので間違えないように。
また、marker nameの行があるかないか、個体番号、個体群番号の列があるかないかの指定も行う。
input fileでalleleを横並びでつくった場合は、ここでデータ形式を指定する。
正常にinput fileが読み込まれると、以下のような画面になる。
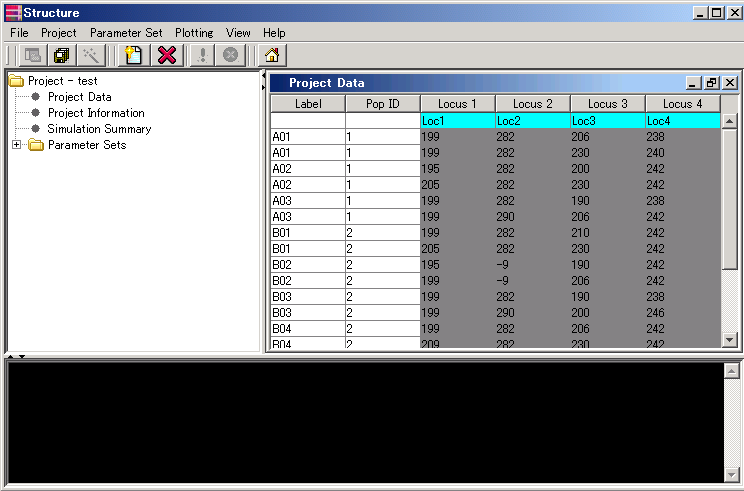
(これはAlleleが縦並びの場合。)
[Parameter Set]→[New]を開き、解析内容などの設定を行う。
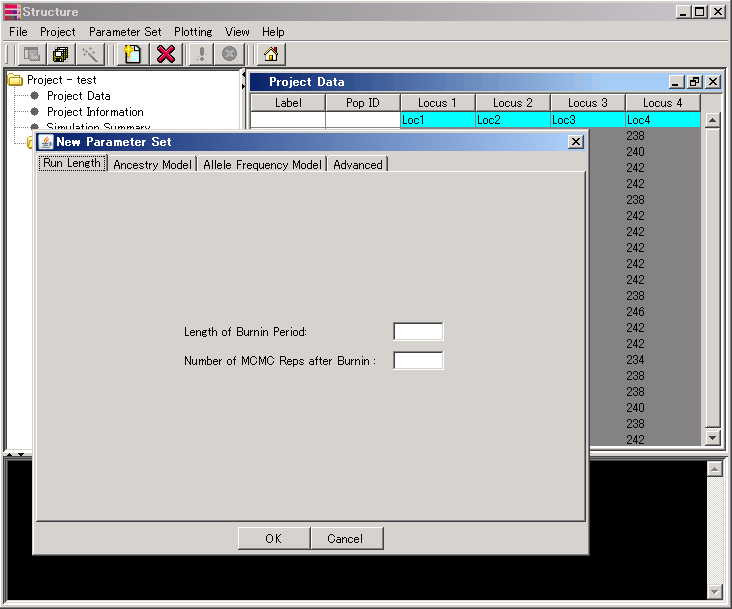
まず[Run Length]では、Burn-inとMCMCの繰り返し回数を指定する。
詳しいことは置いといて、両方とも10000〜100000が推奨されてる。
あとのタブ[Ancestry Model]〜[Advanced]は、詳しいことはちょっとわからないけど、普通(?)はデフォルトのままでいいと思う。
全て指定したら[OK]を押して、parameterの名前を決めると設定は終わり。
次に、[Project]→[Start a job]から[Structure Scheduler]を表示。
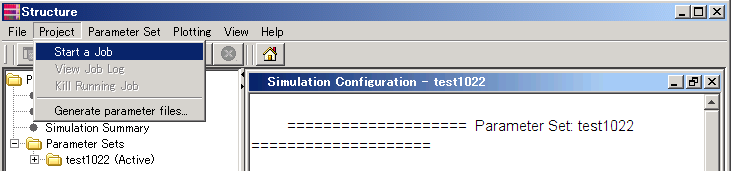
ここで、K(クラスター数)や、各Kあたりの繰り返し数を決める。(だいたい10〜20かな)
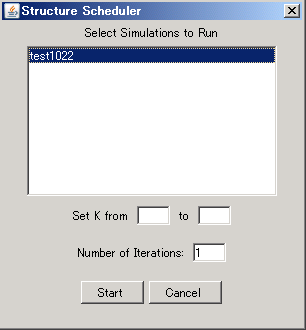
たとえばここで、
Set K from 1 to 8
Number of Iterations: 10
とすると、
K=1について10回、K=2について10回、…、K=8について10回
の、計80回のシミュレーションが行われる。
これは個体数やKが増えるにしたがって、通常、はかりしれない時間を要するので、帰宅前にセットしておけばいいだろう。
STRUCTUREの計算中はパソコンが非常に重くなるので、他の作業はしんどいと思う。
翌朝に解析が終わっていれば各シミュレーションごとに結果が出力される。
以下は、ある学生の結果の例。個体群14、K=1〜14、繰り返し10回で試行。
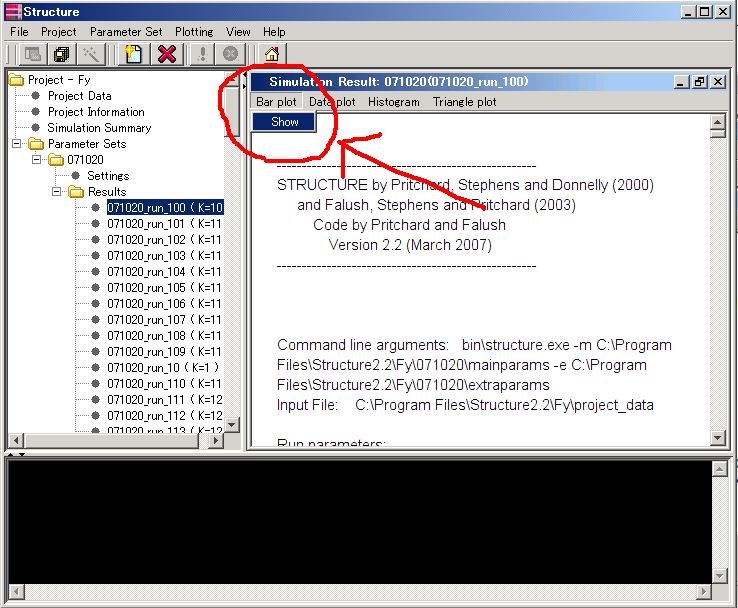
結果の色分けされた図は、Resultのひとつを選択し、右の窓に表示された[Simulation Result]の[Bar plot]→[show]から確認できる。
ここは、結構見逃しやすい。
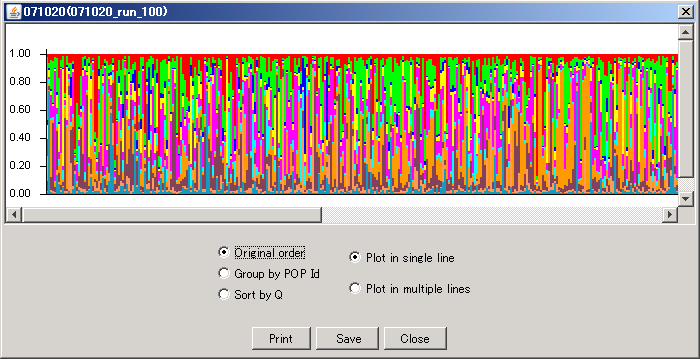
ここで、もし個体群番号を指定しているなら、[Group by POP Id]をチェックすると個体群ごとに仕切られて表示される。
Kの推定
解析終了後の画面から、[Simulation Summary]を表示する。
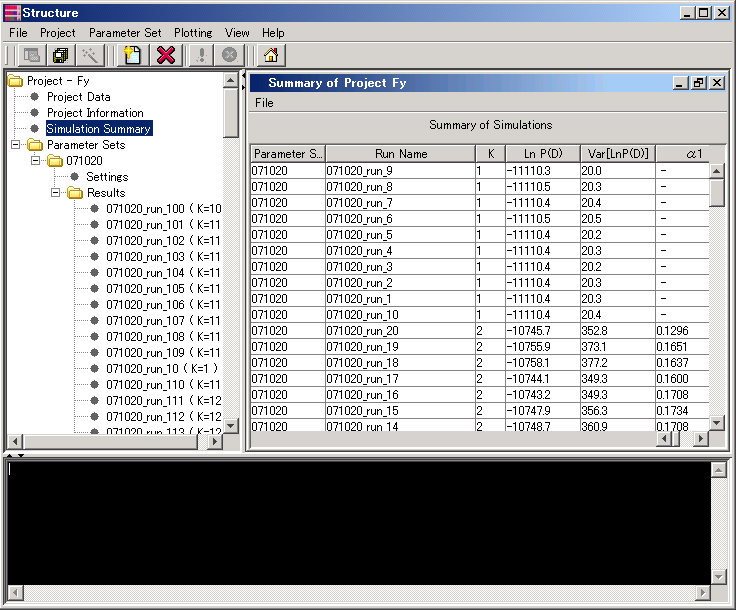
ここから、各Kでの対数尤度Ln P(D)の平均値を求め、Ln P(D)が最大値をとるときのKを採用する。
ただし同じKでLn P(D)にばらつきが大きければ、⊿Kという指標を用いた方がいい。(Evanno et al. 2005)
ここで、一番上のダウンロードファイルを使えば、比較的ラクに⊿Kを求められる。